LOS PLÁSMIDOS Y SU ROL EN LA TRANSFERENCIA HORIZONTAL DE GENES
Lucas Castellani, Ileana Salto, Gonzalo Torres Tejerizo, Mariano Pistorio
Los plásmidos son elementos más pequeños que los cromosomas bacterianos y contienen en su estructura molecular genes que generealmente no se encuentran en el cromosoma. Si bien estos elementos contribuyen a la diversidad y plasticidad genética de los microorganismos, no son, en principio, esenciales para la vida microbiana, y por lo tanto, constituyen un recurso genético distinto, dinámico y adicional a los genes cromosomales. La importancia de los plásmidos se debe a su papel preponderante en la transferencia horizontal de genes dentro de las poblaciones bacterianas y como herramientas para la manipulación genética.
Los plásmidos permiten a las poblaciones bacterianas “muestrear” el pool de elementos genéticos móviles (moviloma) buscando así rasgos adaptativos que puedan ser ventajosos para la supervivencia bajo presiones selectivas locales, como resistencia a antibióticos, metales y contaminantes orgánicos del ambiente o la capacidad de utilizar fuentes de carbono alternativas. En el laboratorio estamos interesados en el aislamiento, secuenciamiento y caracterización funcional de los recursos genéticos asociados a plásmidos presentes en la comunidad bacteriana relacionada a ambientes tan distintos como los ambientes agrícola y hospitalario.
El análisis detallado de los plásmidos representa una poderosa herramienta para avanzar en el conocimiento de sus secuencias génicas, en particular nos permitirá el estudio de funciones adaptativas y/o de relevancia biotecnológica portadas en los mismos.
De esta manera se espera obtener nuevos datos sobre qué tipo de plásmidos se selecciona (grupo de incompatibiliad/tipo de origen de replicación) en cada uno de los ambientes y las relaciones que existen entre los distintos ambientes (interrelación/propagación de la información).
METAGENÓMICA APLICADA AL ESTUDIO DE MICROBIOMAS DE INTERES AGRICOLA. EFECTO DE LA BIOGEOGRAFÍA SOBRE EL MICROBIOMA DE LA VID
Mónica Oyuela Aguilar, Dr. Mariano Pistorio
En el marco de la agricultura sustentable, existe un creciente reconocimiento de la importancia económica y biológica de los microorganismos del suelo. La productividad de los suelos agrícolas depende en gran medida de distintos procesos llevados a cabo por las comunidades microbianas presentes en el suelo. En la vitivinicultura esta comunidad microorganismos compleja y no del todo caracterizada juega un papel clave en las distintas etapas y en procesos de vinificación, desde ayudar a las plantas en el acceso de nutrientes del suelo y contribuir a la sanidad de la planta a través de protección contra patógenos, hasta el proceso de fermentación que transforma el mosto en vino con su complejo conjunto de aromas y sabores. El conocimiento que se dispone actualmente sobre la estructura de comunidades microbianas está basado en aquellos microorganismos cuyo crecimiento se ha logrado en condiciones de laboratorio. El análisis metagenómico consistente en la investigación de las comunidades microbianas a partir de ADN extraído directamente de muestras ambientales, permite encarar el estudio y la descripción de la complejidad microbiana de suelo independientemente del cultivo de la misma. El plan propuesto fue concebido bajo la hipótesis de que gracias a los recientes desarrollos en el estudio de la microbiología, principalmente debido a las nuevas técnicas de secuenciación, se presenta una buena y única oportunidad para utilizar técnicas de laboratorio y bioinformáticas metagenómicas modernas con el fin de obtener una gran comprensión de los complejos procesos a través de los cuales los microbios se asocian con las vides y en consecuencia posteriormente afectan los vinos.
HERRAMIENTAS MICROBIOLÓGICAS, MOLECULARES Y ÓMICAS APLICADAS AL ESTUDIO DE LA INTERACCIÓN SIMBIÓTICA ENTRE RIZOBIOS Y LEGUMINOSAS. CARACTERIZACIÓN DE LAS PROPIEDADES SIMBIÓTICAS Y DE VIDA LIBRE DE Rizobium favelukesii, NODULADOR DE ALFALFA Y OTRAS LEGUMINOSAS
Juliet Nilsson, Gonzalo Torres Tejerizo, Mariano Pistorio
En el marco de una agricultura sustentable, la Fijación Biológica de Nitrógeno (FBN) realizada por bacterias en asociación simbiótica con leguminosas, es un factor importante desde el punto de vista de la conservación de suelos y de la productividad agropecuaria, siendo la misma un evento crucial para asegurar la entrada de nitrógeno atmosférico a los suelos agrícolas. La alfalfa es la leguminosa forrajera más cultivada para la alimentación animal y su asociación simbiótica con el rizobio eficiente, ya sea autóctono y/o inoculado, se ve afectada en suelos con pHs moderadamente bajos. Hace algunos años se aislaron, de suelos del centro de Argentina, un grupo de rizobios denominados Rizobium favelukesii, tolerantes a la acidez y muy competitivos para la nodulación de alfalfa pero ineficientes en la FBN, presentando así características más parasíticas que simbióticas, que lo convierte en un potencial factor de riesgo al competir con el rizobio eficiente Ensifer (Sinorhizobium) meliloti.
La caracterización los Rhizobium favelukesii contribuira a entender aspectos básicos de la biología de los mismos, como la tolerancia a la acidez o mecanismos implicados en la fijación biológica de nitrógeno aún no descriptos. Así, en un futuro cercano podremos evaluar la posibilidad de: a) obtener en el entorno genético de los rizobios tipo Oregon una fijación biológica de nitrógeno eficiente, y/o b) obtener variantes del simbiote eficiente E. meliloti que sean capaces de tolerar condiciones ácidas. Ambas opciones tienen como finalidad aumentar la producción de alfalfa en los suelos agrícolas de Argentina.
TRANSDUCCIÓN DE SEÑALES EN BACTERIAS
Carolina Vacca, Agustina Pérez, Juliana Heredia, Keila Belhart y María Florencia Del Papa
En el marco de una agricultura sustentable la Fijación Biológica de Nitrógeno por rizobios en asociación simbiótica con leguminosas es un factor importante desde el punto de vista de la conservación de los suelos y la productividad agropecuaria. Debido a esto, el estudio de las asociaciones simbióticas rizobio-leguminosa ha sido muy activo, aumentando en forma considerable las posibilidades actuales de generar bacterias transgénicas con capacidades simbióticas superiores. Contrapuesto a los avances en este sentido, es poco lo que se conoce en relación a los determinantes genéticos involucrados en la tolerancia al estrés.
Desde el punto de vista práctico, el conocimiento de los mecanismos y procesos asociados con la tolerancia a un estrés será determinante para el diseño de estrategias que permitan mejorar las bacterias utilizadas para inocular un determinado suelo desfavorable.
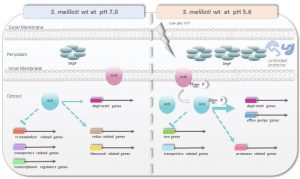
En el grupo estamos concentrados en el estudio de señales externas que modulan la actividad biológica de sistemas de regulación transcripcional, y la caracterización funcional de estos sistemas y de los genes bajo su control, tomando la asociación Sinorhizobium meliloti-alfalfa como sistema modelo. Nuestros estudios involucran el uso de estrategias bioquímicas, genéticas, y de biología molecular, incluyendo el uso de microscopía y análisis ómicos. Nos proponemos además concebir herramientas con potencial aplicación del conocimiento generado a la implementación y mejora de bioproductos con utilidad agro/tecnológica.
CARCATERIZACIÓN GENÓMICA Y FUNCIONAL DE PLÁSMIDOS AISLADOS DEL AMBIENTE
Antonio Lagares, Mariano Pistorio, María Florencia Del Papa
Empleamos una aproximación genómica (basada en secuenciamiento masivo y bioinformática) y funcional para la exploración y recuperación de actividades biológicas plasmídicas de microorganismos a partir de comunidades microbianas sometidas a presiones de selección específicas vinculadas a la presencia de compuestos recalcitrantes. Las mismas podrían ser aplicables a procesos de bioremediación necesarios por el uso de determinadas prácticas agrícolas.
Así también el conocimiento de los plásmidos de dicho ambiente nos permitirá avanzar en el estudio de funciones directamente asociadas a los mismos como la replicación, estabilización, partición y movilización. El conocimiento de la información contenida en estos plásmidos podrá facilitar la predicción de la secuencia de proteínas con potencial uso en aplicaciones biotecnológicas o terapéuticas y enzimas con potencial aplicación en el catabolismo de compuestos recalcitrantes xenobióticos, como pesticidas y herbicidas o de potencial interés en procesos tecnológicos/industriales.
CARACTERIZACIÓN SIMBIÓTICA Y MOLECULAR LOS RIZOBIOS NODULADORES DE Desmodium incanum EN ECOSISTEMAS DE ARGENTINA
María Antonieta Toniutti, María Florencia Del Papa
Nuestro grupo está interesado en caracterizar a nivel bioquímico y molecular los rizobios noduladores de Desmodium incanum como así también la asociación simbiótica entre ambos. Nos proponemos estudiar aspectos fisiológicos, bioquímicos y moleculares de las cepas aisladas de nódulos de Desmodium incanum recolectados en el campo y provenientes de ensayos de laboratorio.
Los estudios para avanzar en la estimación de la competitividad y de la capacidad de fijar nitrógeno de estos rizobios, colaborarán en la tarea de seleccionar cepas más eficientes con potencial interés agronómico.
CARACTERIZACIÓN MOLECULAR Y FENOTÍPICA DEL MICROBIOMA BACTERIANO CULTIVABLE ASOCIADO A SEMILLAS Y PLANTAS DE ALFALFA
José Luis López, Antonio Lagares
Las evidencias actuales muestran a las plantas como organismos naturalmente muy colonizados, tanto en su exterior (rizósferas y filósferas) como en su interior, por una diversidad muy grande de microorganismos. Si bien la mayoría de ellos actúan como comensales sin ningún efecto conocido sobre las plantas que colonizan, múltiples bacterias y hongos establecen con sus hospedantes relaciones ya sea mutualistas o de carácter patogénico. En dicho contexto, el resultado del comportamiento del sistema en un ambiente particular (fitness) resulta de la interacción del conjunto de los genes del huésped y los de todo su microbioma asociado (i.e. el hologenoma). De este modo, la unidad de selección a lo largo del tiempo está constituida por la propia planta y por todo su microbioma (i.e. el holobionte). Un aspecto particularmente importante de las asociaciones entre plantas y microorganismos es el relacionado con la heredabilidad y la dinámica de los microbiomas. En relación con este punto, los microorganismos que colonizan semillas y raíces han sido los que han recibido la mayor atención con información que apoya la existencia de diferentes mecanismos de transmisión que incluyen la via sistémica (in planta), horizontal (entre plantas) y aun vertical de los microbiomas (a lo largo de generaciones). Bajo este modelo la carga microbiana endofítica de semillas sanas parece ser al menos parcialmente heredada a través de generaciones, representando un inóculo inicial importante de las plántulas nacientes que es luego competido por miembros de la comunidad del suelo que arriban más tardíamente. Todas estas consideraciones posicionan al estudio de los microbiomas vegetales como un objetivo central para comprender de modo más acabado las respuestas y evolución de las plantas en su ambiente natural, así como para (luego) hacer un uso práctico racional de las actividades benéficas presentes. Resultados recientes de nuestro grupo orientados a caracterizar la población endofítica de semillas de alfalfa esterilizadas en superficie, revelaron la presencia de bacterias de más de 40 géneros diferentes pertenecientes a 4 phylas distintos que incluyen Proteobacteria, Actinobacteria, Firmicutes y Bacteroidetes. La posición taxonómica de dichos aislamientos fue inferida por una combinación de secuenciamiento parcial de regiones del rDNA 16S y del uso de tecnología MALDI-TOF BiotyperTM Brucker. Entre los aislamientos que estuvieron presentes sin excepción en al menos 3 de las 6 variedades de alfalfa analizadas encontramos miembros de los géneros Arseniciccocus, Arthrobacter, Bacillus, Kocuria, Microbacterium, Microccocus y Paenibacillus, varios de ellos con un alto grado de polimorfismo aun entre aislamientos diferentes de un mismo género. Con un grado menor de ubicuidad, otro género muy frecuente correspondió a varios aislamientos de Pantoea sp. que se caracterizaron por estar presente en números elevados comparados con los correspondientes al resto de la mayoría de los endófitos de semilla. La colección de más de 240 aislamientos endofíticos de alfalfa se encuentra preservada a -80°C como parte del cepario de nuestro grupo en el IBBM. En clones representantes de los diferentes géneros que colonizan alfalfa hemos comenzado a investigar características fenotípicas asociadas a la promoción del crecimiento vegetal y al biocontrol, y a definir sistemas modelo para el estudio de los determinantes genéticos que son requeridos para la colonización endofítica de raíces y de la parte aérea de las plantas hospedadoras.
BÚSQUEDA, IDENTIFICACIÓN Y CARACTERIZACIÓN FUNCIONAL -A ESCALA GENÓMICA- DE DETERMINANTES GENÉTICOS DE RIZOBIOS REQUERIDOS PARA LA COLONIZACIÓN DE LA RIZÓSFERA DE PLANTAS HOSPEDADORAS Y NO-HOSPEDADORAS
María Eugenia Salas, Maricio Javier Lozano, Antonio Lagares
Los rizobios son α- y β-proteobacterias habitantes del suelo que poseen la capacidad de asociarse en simbiosis con raíces de plantas leguminosas, para formar nódulos radiculares fijadores de nitrógeno atmosférico. Dicha asociación, que resulta de una compleja serie de eventos coordinados de comunicación y diferenciación de ambos simbiontes, resulta hoy un modelo de referencia para el estudio de diversas interacciones entre plantas y organismos procariotes tanto simbióticos como patogénicos. Rizobios y leguminosas se asocian a través de un complejo proceso de interacción en el que la percepción recíproca a lo largo del camino simbiótico, sirve de señal de diferenciación para inducir cambios bioquímicos y morfológicos de ambos participantes.
En ese proceso, el número de nódulos finales está cuidadosamente regulado para evitar infecciones innecesarias que son energéticamente costosas para la planta. Así, dado que el nicho simbiótico está naturalmente limitado, acceden al mismo aquellas cepas del suelo que muestran las mejores cualidades en términos de capacidad asociativa (mayor competitividad simbiótica). Las evidencias actuales indican que la competitividad es un fenómeno complejo, en el que son particularmente relevantes los procesos que tienen lugar en la vida rizosférica temprana (generar y tolerar toxinas, capacidades metabólicas para el uso de compuestos de origen radicular, tolerancia a estreses, y movilidad, entre otros).
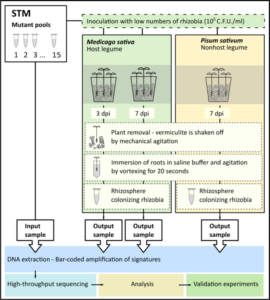
Lamentablemente, y a pesar del detallado conocimiento actual del proceso de infección y desarrollo de nódulos por los rizobios, poco se conoce sobre los genes asociados a la colonización rizosférica. Usando técnicas modernas de selección fenotípica asistida por mutantes etiquetados (STM, Signature-Tagged Mutagenesis) en nuestro laboratorio hemos enfocado la búsqueda, identificación y caracterización de los determinantes genéticos de rizobios asociados a la colonización temprana de la rizósfera de plantas hospedadoras y no-hospedadoras.
El avance espectacular de los últimos años en la disección del proceso simbiótico, y la disponibilidad de tecnologías de alta penetración (mutagénesis etiquetada a escala ómica, el secuenciamiento profundo) nos acercan cada vez más a la posibilidad incipiente de abordar el mejoramiento racional del sistema, expandiendo la potencialidad de los procesos clásicos de selección empírica de nuevas cepas. Es en ese marco que el proyecto enfoca la búsqueda e identificación de genes simbióticos asociados a una al proceso de abordaje inicial a la planta por parte de los rizobios. La identificación de marcadores asociados a los estadíos tempranos de comunicación rizobio-raíz, y
el análisis de las consecuencias funcionales de su pérdida/sobre-expresión; son aspectos que necesitan ser aun consolidados para determinar de manera más acabada cuáles son las instancias en las que el proceso simbiótico se limita y (por tanto) puede ser facilitado por manipulación de los participantes.
IDENTIFICACIÓN Y CARACTERIZACIÓN DE MARCADORES GENÉTICOS DE RIZOBIOS ASOCIADOS A LA PERSISTENCIA DE LAS BACTERIAS EN SOPORTES SÓLIDOS (TURBA) Y LÍQUIDOS DE FORMULACIONES COMERCIALES DE INOCULANTES
María Eugenia Salas, Maricio Javier Lozano, Antonio Lagares
Los avances del conocimiento y la tecnología en la últimas tres décadas han revolucionado las estrategias de producción agrícola hacia rindes cada vez más elevados, con mayor cuidado de la fertilidad del suelo, y con empleo cada vez más dominante de agentes biológicos promotores del crecimiento (microorganismos promotores del crecimiento y la sanidad vegetal: PGPB, Plant Growth Promoting Bacteria). El avance creciente en el uso de bioproductos frente a la aplicación de agroquímicos, se sustenta en tres pilares básicos que a saber son: a) su seguridad (recrean interacciones naturalmente existentes, sosteniendo asociaciones y procesos bio-geoquímicos del ecosistema), b) su eficiencia, y c) su menor costo de producción. El empleo de bioproductos camina así de modo firme hacia el uso de diversos microorganismos con efectos benéficos específicos sobre las plantas. En el contexto precedente, los biofertilizantes elaborados en base a rizobios (“inoculantes”) en particular representan los productos de uso más extendido, de mayor impacto eco-agrícola y económico, y sobre los que más esfuerzo tecnológico se invertido. La razón de ello radica en que son fuente de fijación biológica de nitrógeno al suelo, con experiencia de uso de más de un siglo. Sin embargo, la inoculación con rizobios seleccionados en base a su calidad fijadora de nitrógeno no siempre impacta del modo esperado en los sistemas de producción a campo. Es un hecho frecuente observar la pérdida de viabilidad microbiana entre la producción y el uso del biofertilizante a campo, y también falta de competencia de las cepas introducidas frente a las presenten en el suelo (pueden ser menos eficientes para fijar nitrógeno). Resulta por tanto importante mejorar la calidad de los inoculantes en base a conocimiento que permita delinear estrategias que mejoren la supervivencia de los rizobios en los productos comerciales, y que promuevan la competencia simbiótica temprana frente a la raíz en crecimiento de la semilla recién germinada. Utilizando tecnologías de STM (Signature Tagged Mutagenesis) y herramientas bioinformáticas, en nuestro laboratorio hemos iniciado la búsqueda de genes de los rizobios asociados a la expresión de una buena sobrevivencia en soportes inoculantes, empleando la bacteria modelo Sinorhizobium meliloti simbionte de alfalfa. Una aproximación como la anterior permitirá analizar la presencia de genes homólogos en otros rizobios y evaluar la validez de los resultados en otros sistemas simbióticos de importancia agrícola (soja, poroto, otros). Desde lo práctico, trabajamos para contar con nuevos marcadores cuya expresión se asocie a mejoras en las características de persistencia en la fase de almacenamiento, hacia el diseño de nuevos protocolos para trazabilidad de calidad de rizobios en formulaciones inoculantes.
ABORDAJE AL AISLAMIENTO, SECUENCIACIÓN Y CARACTERIZACIÓN FUNCIONAL DE MOVILOMAS PLASMÍDICOS EN UN RIZOBIO MODELO Y SUS BACTERIAS SIMPÁTRICAS. CONTENIDO INFORMATIVO, TRANSFERENCIA HORIZONTAL DE GENES Y RELACIONES CON LOS REPLICONES CELULARES MÁS ANCESTRALES (CROMOSOMA, MEGAPLÁSMIDOS)
José Luis López, Antonio Lagares
Como consecuencia de su forma de vida e historia evolutiva, la fisiología y las respuestas bioquímicas de los organismos unicelulares son esencialmente diferentes de las expresadas por las células eucarióticas en organismos multicelulares. Mientras los organismos unicelulares se hallan generalmente en ambientes diversos y con condiciones cambiantes, las células que forman parte de organismos multicelulares han evolucionado para ejercer funciones más especializadas gracias a la protección que reciben desde redes homeostáticas que amortiguan las variaciones del entorno. En este contexto, las bacterias han concentrado eficientemente en sus pequeños genomas la información necesaria para afrontar los diferentes desafíos que le imponen los cambios ambientales (estreses abióticos y bióticos). En términos genéticos (informativos), las bacterias han concentrado la mayoría de sus funciones basales (housekeeping) en sus replicones más grandes (los cromosomas) dedicando compartimientos genéticos especiales y móviles (plásmidos, islas de ADN, transposones, integrones) para hacer frente a respuestas adaptativas ante condiciones de estrés y menos ubicuas en sus ciclos de vida. La partición del material genético en módulos móviles, como elementos funcionales accesorios al cromosoma (“moviloma”, mobilome), facilitó la reducción del tamaño de los genomas permitiendo compartir genes (funciones) transitoriamente importantes (adaptativas) entre los diferentes miembros de la comunidad bacteriana. Laa partición genética con redundancia previene, al mismo tiempo, la pérdida masiva de información cuando miembros individuales de la comunidad bacteriana mueren. El conjunto de plásmidos y otros elementos móviles en un ambiente dado representa un recurso genético donde las bacterias codifican, moldean, ensayan y concentran información para hacer frente a los desafios que imponen los cambios del medio extracelular. En nuestro laboratorio trabajamos, desde hace más de 10 años, en la caracterización clásica y del contenido informativo del moviloma plasmídico de Sinorhizobium meliloti. Los resultados nos han permitido conocer el grado de movilidad horizontal via conjugación expresada por los plásmidos, caracterizar sus sistemas de replicación y movilización, y contar nueva información del compartimento plasmídico a escala de megabases. A partir de dicha información estamos actualmente: a) analizando el tipo de genes presentes en los plásmidos crípticos (funciones), b) explorado relaciones de ortología con plásmidos (que ya hemos aislado y secuenciado) de otras bacterias colonizadoras de la raíz de alfalfa (bacterias simpátricas) como signo de intercambio génico intesgenérico/interespecífico, y c) explorando relaciones entre la información contenida en los plásmidos crípticos respecto de la presente en el replicón cromosomal y en los dos megaplásmidos simbióticos (pSyma y pSymb), hacia la elaboración de un modelo que explique la dinámica del flujo génico y la plasticidad genómica en un rizobio modelo.
ESTUDIOS FUNCIONALES Y ECOLÓGICOS DE MICROORGANISMOS DEL SUELO PARA UNA PRODUCCIÓN AGRÍCOLA SUSTENTABLE
Walter Draghi, Antonio Lagares
La sustentabilidad de los sistemas de producción agrícola es indispensable para mantener la productividad de los recursos naturales. En este sentido el uso de microrganismos es una técnica ampliamente utilizada que provee de ventajas competitivas a los cultivos frente a condiciones ambientales adversas que pueden presentarse en las diferentes fases de crecimiento de los mismos.
Para ello estudiamos el rol de diversos microrganismos rizosféricos que promueven el crecimiento vegetal a través de diversos mecanismos, como lo son la fijación biológica de nitrógeno, o la producción de fitohormonas, entre otros. El plan de trabajo que desarrollamos se basa en dos ejes principales:
1- Por un lado hemos utilizado acercamientos experimentales orientados a avanzar en la obtención de cepas inoculantes más persistentes y eficientes para la simbiosis y fijación de nitrógeno en suelos ácidos. Para ello se estudian las bases bioquímicas y genéticas de la adaptación y tolerancia a la acidez en Sinorhizobium meliloti, simbionte de alfalfa, a través de la aplicación de técnicas de expresión global (ómicas). También se aborda el estudio para la generación de fenotipos celulares resistentes a la acidez (ATR), los cuales han demostrado una mayor capacidad de infección en plantas, para poder generar simbiontes más infectivos en ambientes bajo estrés acido.
2- Por otro lado abordamos el estudio de la ecología poblacional de especies pertenecientes al género Burkholderia spp. presentes en suelos agrícolas de nuestro país. Dichas poblaciones han demostrado poseer diversas características promotoras del crecimiento vegetal, las cuales las transforman en blanco potenciales para su uso en formulaciones comerciales. Utilizando técnicas microbiológicas (clásicas y moleculares) estudiamos la estructura poblacional de este género bacteriano y su variabilidad respecto de las prácticas agrícolas utilizadas, las cuales pueden alterar la diversidad funcional de estas cepas de dicha especie asociadas a cultivos de interés agrícola.
BÚSQUEDA DE DETERMINANTES INVOLUCRADOS EN LA DIFERENCIACIÓN DE LOS BACTEROIDES EN LA SIMBIOSIS MEDICAGO-RHIZOBIUM
Gonzalo Torres Tejerizo, Lucas Castellani, Abril Luchetti
La simbiosis entre Medicago-rhizobia es un proceso altamente complejo. El desarrollo de esta asociación se lleva a cabo bajo la expresión coordinada de varios genes relacionados con la simbiosis, tanto en la planta como en la bacteria. El proceso general que conlleva a la formación de nódulos fijadores de N, incluye numerosos pasos, como el desarrollo de hilos de infección, la invasión de células corticales, la endocitosis del rizobio y la diferenciación del bacteroide. Estos bacteroides son los responsables de una exitosa fijación de N. Ha sido descripto que péptidos ricos en cisteínas específicos de nódulos son esenciales para la diferenciación del bacteroide. Estos péptidos son producidos por la planta y dirigidos a la bacteria. Sin embargo, todavía se desconocen otros genes involucrados en esta comunicación tardía y los mecanismos moleculares que ocurren durante dicho proceso. Este trabajo se enfoca en desentrañar los determinantes implicados en la diferenciación de los bacteroides en los nódulos, con el objetivo de comprender mejor el proceso y optimizar nuestras habilidades para maximizar la producción de alfalfa. Para ello, se realizaron aproximaciones transcriptómicas (RNAseq) para analizar la expresión de genes tanto de la planta como de la bacteria, utilizando como modelo rizobios eficientes e ineficientes en la fijación de nitrógeno.
EXPLORACIÓN DE COMPUESTOS ANTIMICROBIANOS CON ACTIVIDAD ANTIMICOBACTERIANA
María Carla Martini, Ludmila Careri
La tuberculosis se encuentra entre las diez principales causas de muerte a nivel mundial, lo que la convierte en un tema prioritario de salud a nivel global. Tan sólo en el año 2021 se reportaron 10 millones de casos de tuberculosis y 1,6 millones de personas perdieron la vida por esta enfermedad, número que se incrementó por primera vez en años debido a la aparición de la pandemia de SARS-Cov-2. La emergencia de resistencia a antibióticos y la diseminación de cepas resistentes de Mycobacterium tuberculosis, el patógeno responsable de la tuberculosis, es una de las principales causas que limitan el tratamiento y la erradicación de esta enfermedad. Las infecciones causadas por cepas multirresistentes son particularmente difíciles de tratar, ya que el paciente debe ser tratado por 24 meses con drogas alternativas, que son menos eficaces y tienen severos efectos secundarios que impactan drásticamente en su calidad de vida. Por otro lado, otras micobacterias están emergiendo como patógenos humanos, siendo particularmente mortales en pacientes inmunocomprometidos. La resistencia intrínseca de estas especies micobacterianas, sumado a la incompatibilidad de ciertos antibióticos con algunas drogas usadas en pacientes inmunocomprometidos resaltan la necesidad urgente de desarrollar nuevos antibióticos con nuevos mecanismos de acción. En este contexto, esta línea de investigación pretende explorar nuevos compuestos que presenten actividad antitubercular, a fin de mejorar los tratamientos de la tuberculosis multirresistente. En particular, nos enfocamos en los péptidos antimicrobianos (AMPs) con actividad antimicobacteriana producidos por otras bacterias.